La technique PCR : Différence entre versions
(Page créée avec « '''La PCR (Polymerase Chain Reaction) est une technique d'amplification génétique in vitro qui a été conçue au début des années 80 par un chercheur américain, Kary Mu… ») |
(Aucune différence)
|
Version du 10 juillet 2014 à 08:16
La PCR (Polymerase Chain Reaction) est une technique d'amplification génétique in vitro qui a été conçue au début des années 80 par un chercheur américain, Kary Mullis, travaillant au sein d’une firme biotechnologique californienne. Cette technique, qui a révolutionné les approches expérimentales en biologie moléculaire, a été publiée pour la première fois en 1985 dans la revue scientifique "Science". En 1993, Kary Mullis recevra le Prix Nobel de Chimie pour sa découverte de la PCR. Comme tout développement technologique nouveau, la PCR a d'abord été investiguée dans les domaines humains et vétérinaires. A partir des années 90, elle commence à être utilisée dans le domaine agricole. D'abord en tant qu'outil d'investigation pour la recherche fondamentale : étude et compréhension des génomes des plantes et organismes pathogènes, caractéristiques génétiques des variétés végétales. Puis en tant qu'outil dans le cadre des biotechnologies végétales de transformation (organismes génétiquement modifiés) et de sélection variétale. Enfin, dans le domaine du phyto-diagnostic (détection, identification et caractérisation des agents pathogènes des plantes), où sa puissance en termes de précision et de capacité de détection a permis la mise en place et le développement de tests extrêmement performants. Cet article de l’AgroReporter décrit la technique PCR et ses applications en agriculture.
Principe de la PCR
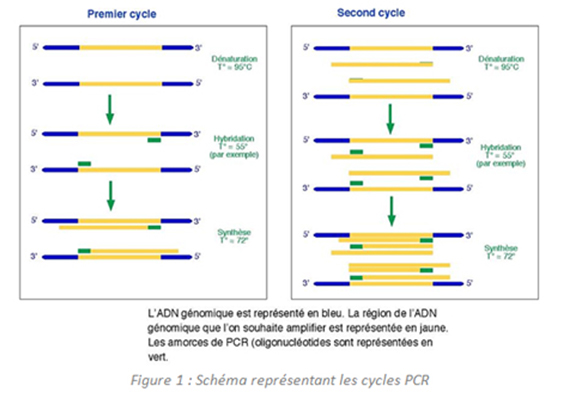
La PCR est basée sur le mécanisme de réplication de l'ADN (1) in vivo : l'ADN bicaténaire est déroulé en ADN monocaténaire, puis dupliqué et ré-enroulé, selon des cycles répétitifs comprenant les trois étapes suivantes :
• Dénaturation de l'ADN par fusion à haute température pour convertir l'ADN bicaténaire en ADN monocaténaire. Cette étape est réalisée à une température comprise entre 93 et 96°C.
• Hybridation à l'ADN cible de deux oligonucléotides utilisés comme amorces. Cette hybridation a lieu à une température comprise entre 55 et 65°C.
• Extension de la chaine d'ADN par addition de nucléotides à partir des amorces en utilisant l'ADN polymérase (2) comme catalyseur en présence d'ions Mg2+. La température optimale de travail de l'ADN polymérase est de 72°C.
- Trois étapes = Un cycle...
Les oligonucléotides sont de courtes séquences d'ADN monocaténaire qui sont différentes les unes des autres et complémentaires des sites de reconnaissance encadrant la séquence d'ADN à amplifier. Les étapes de dénaturation de la matrice d'ADN, d'hybridation des amorces et d'extension des amorces en brins complémentaires constituent un cycle dans la méthode PCR. Après chaque cycle, les brins d'ADN nouvellement synthétisés peuvent servir de matrice dans le cycle suivant. Au fur et à mesure des répétitions de cycles, il s'en suit ainsi une augmentation et une accumulation exponentielle des séquences d'ADN cible représentée dans la Figure 1.
- Une puissance de détection inégalée...
L'amplification, en tant que nombre final de copies de la séquence cible, est exprimée par l'équation suivante :(2n-2n)x
Avec : n = nombre de cycles ; 2n = premier produit obtenu après le premier cycle ; x = nombre de copies de la matrice originelle
Théoriquement, après 20 cycles PCR, en supposant une efficacité de 100% de la PCR, il y aura une amplification d'un facteur de la séquence d'ADN cible initiale, soit 1 048 576 copies de la matrice initiale. On comprend qu'on atteint là des sommets jusqu'alors inégalés en manière de puissance de détection ! Dans la pratique, on considère qu'un protocole PCR de 35 à 40 cycles d'amplification permet d'obtenir entre 100 000 et 1 000 000 de copies d'une séquence d'ADN cible.
- Automatisation
Deux progrès majeurs ont permis aux laboratoires d'automatiser le processus de la PCR :
- Le développement des blocs de température qui peuvent augmenter et abaisser rapidement leur température de matière automatisée et programmée : thermocyleurs ou machines PCR (chauffages et refroidissement par fluides, résistances électriques ou semi-conducteurs)
- Initialement, la méthode PCR utilise une ADN polymérase extraite de la bactérie E. Coli. Toutefois, cette enzyme est rendue inactive par les températures de l'étape de dénaturation, obligeant le rajout d'enzyme "fraîche" pour chaque cycle d'amplification. C'est finalement l'utilisation d'une enzyme thermostable dite "Taq Polymerase (3) , capable de supporter des températures supérieures à 90°C et utilisable en l'état pour la totalité des cycles d'amplification, qui a permis de simplifier et donc d'automatiser la réaction PCR
- PCR en point final / PCR en temps réel
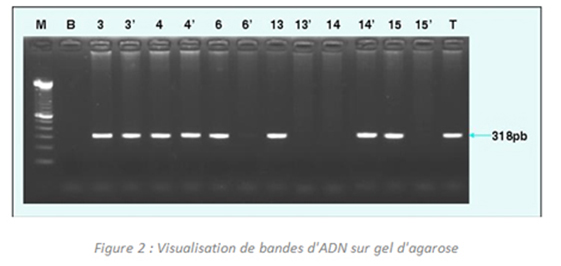
Dans la première génération des tests PCR, dite "PCR Classique" ou "PCR en point final", les produits d'amplification PCR sont analysés au stade terminal du processus analytique. Le contenu des tubes contenant le milieu réactionnel et l'ADN matrice sont déposés sur un gel d'agarose. Si la séquence d'ADN cible a été amplifiée, la quantité d'ADN copiée est en quantité suffisante pour être visualisée, après migration des molécules d'ADN dans un champ électrique, sous la forme d'une fluorescence UV. Cette approche ne permet toutefois d'obtenir que des résultats qualitatifs (réponse présence /absence de la séquence cible).
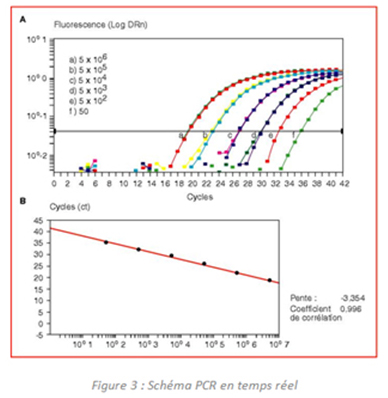
A la fin des années 1990, une deuxième génération de tests PCR fait son apparition sous la forme d'un système qui permet de détecter le produit de la PCR au fur et à mesure qu'il s'accumule. C'est la "PCR en temps réel" dite également "PCR quantitative". L'accumulation des séquences d'ADN cible dans le milieu réactionnel se traduit par une augmentation d'une émission de fluorescence qui est détectée et quantifiée par l'intermédiaire d'une caméra CCD (Charge-Coupled Device). Ce système expérimental permet de suivre en temps réel, cycle par cycle, l'évolution de la réaction PCR. Elle permet en outre, à l'aide de témoins positifs et négatifs adaptés, d'opérer une quantification précise de la quantité d'ADN cible initialement présente dans l'échantillon analysé.
Applications de la PCR dans le domaine agricole
Globalement, tous les pathogènes des plantes (bactéries, champignons, phytoplasmes, virus et viroïdes) sont susceptibles d’être appréhendés en terme de détection par l’intermédiaire de la technique PCR. La contrainte initiale est toutefois qu’il est nécessaire de disposer d’un minimum de connaissance de son génome pour développer les tests PCR. De même, selon la nature des agents pathogènes, le format de test PCR pourra varier sensiblement. Ainsi, pour les bactéries, champignons et phytoplasmes, dont le génome est constitué d’ADN double brin, les étapes de la PCR peuvent être menées directement à partir d’ADN total extrait du matériel végétal soupçonné d’être infecté. Dans le cas des virus et des viroïdes, dont le génome est constitué d’ARN monocaténaire, il est nécessaire d’ajouter une étape préliminaire au test PCR, la Réverse Transcription (RT) : l’ARN simple brin est transformé en ADN double brin par l’intermédiaire d’une enzyme, la Reverse Transcriptase, agissant sur l’ARN total extrait. On parle alors de test de type RT-PCR.
Les applications de la PCR au diagnostic dans le domaine agricole sont nombreuses. Au laboratoire LCA, le développement et l’utilisation en routine des tests PCR pour la détection des agents pathogènes des plantes remonte à une quinzaine d’années. A ce jour, des prestations d’analyses PCR (PCR en point final et PCR en temps réel) sont proposées pour les Phytoplasmes des arbres fruitiers (Enroulement Chlorotique de l’Abricotier, Prolifération du Pommier et Déclin du Poirier), des plantes maraichères (Stolbur) et de la Vigne (Flavescence Dorée, Bois Noir). Des prestations d’analyses PCR en temps réel sont également proposées pour certains Champignons des céréales (Piétin Verse, Fusariose, Septoriose). Au-delà du phyto-diagnostic, la technique PCR commence à être utilisée par certains laboratoires spécialisés pour caractériser la diversité microbienne d’un sol et pour l’analyse fonctionnelle de ses populations. Elle ouvre ainsi une nouvelle voie dans la caractérisation et la compréhension du fonctionnement des sols…